组学科研学习
- 提问:形成自己感兴趣的科研方向,通过搜索阅读文献和听报告,了解这个方向的前沿进展,关注没有回答清楚的问题。对这些问题,你可以提出哪些新的生物假说或模型?
- 研究方案:将研究问题分解为子问题,寻找合作者,设计研究方案,包括使用或开发前沿组学技术产生数据、整合公共组学数据(如GEO、TCGA数据、Allen Brain Atlas)、开发生信方法。
- 回答:实施和推进研究方案,获得并质控组学数据。 定期总结和讨论课题进展、难点、未来计划,从而回答、调整研究问题或提出新问题。
学习资源
- 【张超】Gene-associated domain (GAD)计算和作图代码
- 相关文献:Cell Rep 2020
- 基于Straw计算基因内部相互作用、R-ggplot2作图
- 【刘玉婷】GitHub主页,包括ChIP-seq/RNA-seq/Hi-C数据处理的脚本
- 单细胞多组学分析:流程与R代码,包括预处理、可视化、pseudobulk expression、gene signature、DEG、Monocle2、SCENIC
- 推荐:一个Harvard 课程,包括生物数据中常用的统计分析原理和软件,还有编程基础教学,适合刚入门的同学学习。包括视频和课件。
- 【吕钰麟】一套用于生成ChIP-seq预处理流程脚本的代码
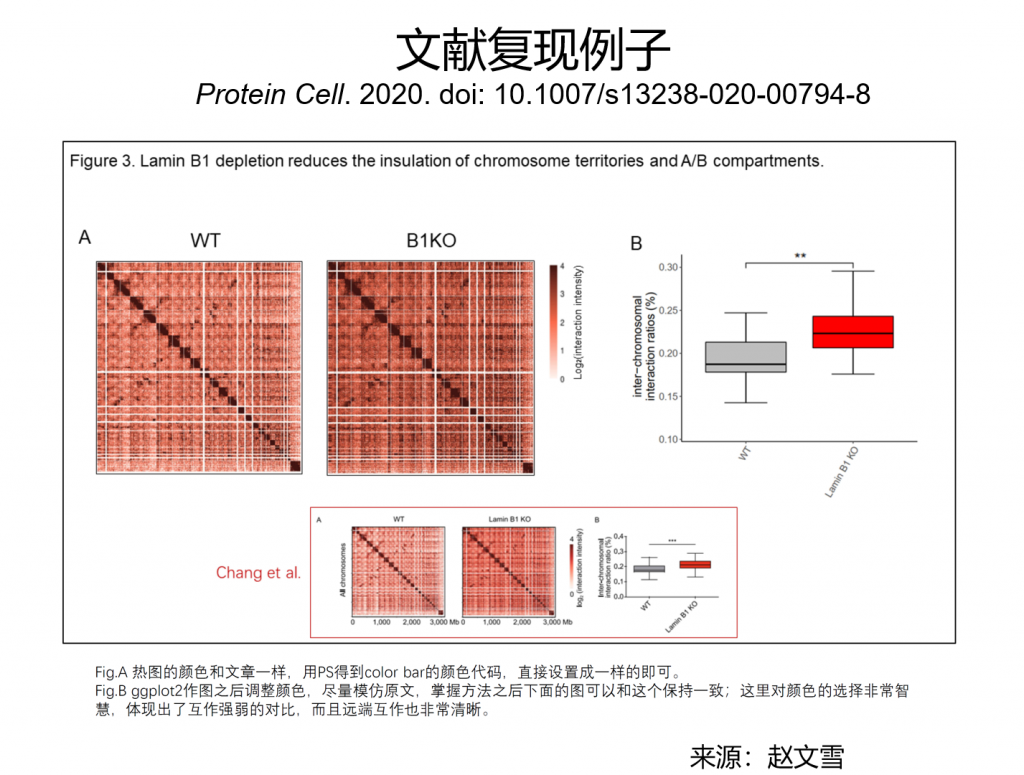
文献复现
从感兴趣的研究方向中搜索前沿文献。选择一篇分析方法有代表性、原始数据可下载的论文(文献选择见下,也可在GEO数据库中用关键词搜索):
- Cell. 2021. 184(3):723-740.e21.(中文介绍)
- Cell Rep. 2020. 32(13):108206.(中文介绍)
- Cell Res. 2022a. 32(1):38-53.(中文介绍,一作历程)
仔细阅读这篇论文的方法和补充材料中的分析方法,从尽可能原始的数据开始做数据处理、分析和做图。目标是自己做出论文中的大部分图表并与它们比较(例子见下图)。